آموزش CytoScape ابزاری برای تحلیل و بصری سازی گراف شبکه
معرفی و آموزش cytoscape
در پست های قبل برنامه cytoscape را با نرم افزار گفی مقایسه (اینجا) کردیم. همچنین نرم افزار گفی را (در اینجا) به طور تقریبا کامل آموزش دادیم در این مبحث میخواهیم به آموزش CytoScape بپردازیم.
سایتواسکیپ یک نرم افزار برای آنالیز و تجسم سازی شبکه های اجتماعی است و در حوزه های مختلفی از جمله بیوانفورماتیک، آنالیز شبکه های اجتماعی و وب معنایی را کاربرد دارد. نسخه جدید این نرم افزار با معماری مدولار است. Cytoscape یک نرم افزار قابل توسعه است. نسخه جدید با معماری مدلاری است. یک کتابخانه جاوا اسکریپت برای آنالیز گراف شبکه است و همچنین یک کامپونت به نام Cytoscape.js برای استفاده در برنامه های تحت وب دارد. اجرای انواع الگوریتم های طرح بندی،سازگار با پلاگین، توانایی فیلتر کردن گره و قابلیت سفارشی کردن صفحه نمایش ،پشتیبانی از زبان های مختلف برای داده ها و جستجوی گره هدف با جستجوی پیشرفته قابل اجرا است. هدف از ایجاد این نرم افزار برای کارهای تجاری و دانشجویی است و نسخه اولیه آن در سال 2002 و نسخه پایدار آن در سال 2015 انتشار پیدا کرده است.
به عنوان یک ابزار رایگان متن باز، Cytoscape به یکی از محبوب ترین ابزار ترسیم و آنالیز شبکه در حیطه زیست شناسی مبدل شده است. به این برنامه سالانه در بیش از پانصد مقاله استناد می شود و ماهیانه بیشتر از هشتصد بار دانلود می شود. در این بسته نرم افزاری ترسیم و آنالیز شبکه های برهمکنش مولکولی و مسیرهای زیستی و همچنین ادغام چنین شبکه هایی با حاشیه نویسی ها و پروفایل های بیانی امکان پذیر می باشد. ویژگی های کاربردی دیگری نیز از طریق 200 افزونه (Plugins) به این بستر نرم افزاری اضافه شده است.
ویژگی های Cytoscape : ابزاری برای تحلیل و بازنمایی تمام انواع شبکههای پیچیده است. نرم افزاری شناخته شده در بیوانفورماتیک. قادر به تحلیل شبکه های ایستا و پویا است.همه نوع شبکهای را تحلیل میکند. در ابتدا برای بیو انفورماتیک طراحی شد اما اکنون در همه زمینه ها کار برد دارد. دارای پلاگینهای مختلفی است. Cytoscape مبتنی بر جاوا است و متن باز میباشد و لایسنس آن (LGPL) است.
موارد استفاده از آن نیز بسیار گستردهتر از ابزارهای دیگر میباشد و با توجه به موارد و کتابهای آموزشی متعدد این ابزار جهت استفاده در پروژه ها پیشنهاد میگردد. برخی از ویژگیهای مهم این ابزار عبارتند از:
- یک کتابخانه جاوا اسکریپت میباشد جهت تجزیه و تحلیل و مجسم سازی گراف (اغلب گرافهای شبکه مانند)
- بهینه شده جهت بازدهی بالا
- عدم وابستگی به دیگر کتابخانهها
- سازگاری با:
- CommonJS/Node.js
- AMD/Require.js
- jQuery
- npm
- bower
- مرورگرهای روز دنیا
- اسناد کافی
- روشهای نمایش متنوع
- پشتیبانی از رخدادها مختلف
- متحرک سازی
- قابلیت اضافه کردن افزونه
نقاط ضعف
- عدم توانایی در چرخاندن زاوید دید دوربین
- پرهزینه بودن رسم گراف
- پرهزینه بودن رسم یال بین نودها
- همچنین بسیاری از عملیاتهای نمایش مانند بزرگنمایی و غیره برای این ابزار پرهزینه میباشد.
موارد استفاده cytoscape
- بیشترین کاربرد سایتواسکیپ در بیولوژیک (biological) و تحلیل شبکه های اجتماعی به چشم می خورد.
برای اطاعات بیشتر به اینجا مراجعه کنید
نصب cytoscape
ابتدا نسخه متناسب با سیستم عامل خود را از آدرس زیر دانلود و نصب کنید. برای اجرای این برنامه باید، نسخه Java8 روی ویندوز نصب باشد. فرایند نصب نکته پیچیده که نیاز به توضح داشته باشد ندارد.
| آدرس اینترنتی: |
http://www.cytoscape.org/ |
منوهای Cytoscape
در این قسمت از آموزش CytoScape به طور خلاصه تمام منوهای موجود در Cytoscape بررسی خواهیم کرد.
منوی File
منوی فایل شامل قابلیت های اساسی کار با فایل ها است:
- File → Open: باز کردن یک فایل
- File → New: برای ایجاد یک شبکه جدید
- File → Save: برای ذخیره وضعیت فعلی گراف
- File → Import : وارد کردن داده ها مانند شبکه ها و ویژگی ها آن
- File → Export : برای خروجی گرفتن از داده ها به صورت تصاویر و فرمت های دیگر
- File → Print: اجازه چاپ را می دهد
- File → Quit : تمام پنجره های Cytoscape را بسته و از برنامه خارج می شود
منوی Edit
منوی ویرایش شامل موارد زیر است:
- عملکرد Undo and Redo
-
گزینه هایی برای ایجاد و نابود کردن دیدگاه ها (بازنمایی های گرافیکی یک شبکه)
-
گزینه هایی برای حذف گره ها و لبه های انتخاب شده از شبکه فعلی.
- Edit → Undo:
- کلیه گره ها و لبه های حذف شده را می توان از طریق Edit → Undo به شبکه بازگرداند.
- Edit → Preferences → Properties : برای ویرایش تنظیمات برای خواص و پلاگین ها
منوی View
منوی View به شما امکان نمایش یا پنهان کردن پنل های زیر را میدهد:
- The network management panel (Control Panel)
- The attribute browser (Data Panel)
- Results Panel
- VizMapper
منوی Select
منوی انتخاب شامل موارد زیر است:
-
گزینه هایی برای انتخاب گره ها و لبه ها
- Select → Use Filters : اجازه می دهد تا فیلتر ها برای انتخاب اتوماتیک بخش هایی از شبکه ایجاد شود که گره ها یا ویژگی های لبه آنها یک معیار فیلترینگ را برآورده می کنند (بخش filters در ادامه را ببینید).
منوی Layout
منوی Layout مجموعه ای از ویژگی های بصری سازماندهی شبکه را دارد:
- چرخش، مقیاس، هماهنگی و توزیع، ابزار برای دستکاری در تجسم سازی شبکه است.
- قسمت پایین منو، انواع الگوریتم های طرح بندی را نشان می دهد که به طور خودکار یک شبکه را بیرون می اندازد.
منوی Plugins.
منوی Plugins حاوی گزینه هایی برای مدیریت پلاگین های شما (نصب / به روز رسانی / حذف) است و ممکن است گزینه های اضافه شده توسط پلاگین هایی که قبلا نصب شده اند، مانند جستجو ادبیات Agilent یا ادغام شبکه ها.
بسته به افزونه های بارگذاری شده، پلاگین هایی که می بینید ممکن است متفاوت از آنچه در اینجا ظاهر می شود متفاوت باشد.
منوی Help
منوی راهنما به شما امکان می دهد تا بیننده را راه اندازی کنید و فهرست مطالب این راهنما را مرور کنید. گزینه “About …” اطلاعاتی درباره نسخه فعلی Cytoscape را نمایش می دهد.
فرمت های فایل شبکه قابل پشتیبانی در Cytoscape :
فرمت SIF فقط گره ها و تعاملات را مشخص می کند، در حالی که فرمت های دیگر اطلاعات بیشتری در مورد طرح شبکه ذخیره می کنند و اجازه می دهد که داده های شبکه را با سایر برنامه های شبکه و منابع داده همگام کنند. به طور معمول، هنگام ساختن شبکه برای اولین بار، فایل های SIF برای وارد کردن تعاملات استفاده می شود، زیرا آنها در ویرایشگر متن یا صفحه گسترده آسان می باشند. هنگامی که تعاملات بارگذاری شده اند و طرح شبکه انجام شده است، شبکه ممکن است به فرمت GML یا XGMML برای تعامل با سایر سیستم ها ذخیره شود. تمام فایل های ذکر شده (به غیر از اکسل) فایل های متنی هستند و شما می توانید آنها را در یک ویرایشگر متنی منظم ویرایش و مشاهده کنید.
- Simple interaction file (SIF or .sif format)
- Nested network format (NNF or .nnf format)
- Graph Markup Language (GML or .gml format)
- XGMML (extensible graph markup and modelling language).
- SBML
- BioPAX
- PSI-MI Level 1 and 2.5
- GraphML
- Delimited text
- Excel Workbook (.xls, .xlsx)
- Cytoscape.js JSON
- Cytoscape CX
توضیحات این فرمتها را در لینک زیر میتوانید مطالعه کنید.
http://manual.cytoscape.org/en/stable/Supported_Network_File_Formats.html
دارای پشتیبانی درون ساز برای فایل های مایکروسافت اکسل (.xls، .xlsx) و فایل های متن دلخواه است. جداول این فایل ها می توانند اطلاعات شبکه و ستون های لبه را داشته باشند. کاربران می توانند ستون ها را با گره های منبع (source)، گره های هدف (target)، انواع تعاملات (interaction) و ستون های لبه (edge column) در هنگام وارد کردن فایل مشخص کنند. برخی از ابزارهای تجزیه و تحلیل شبکه دیگر، مانند igraph (http://cneurocvs.rmki.kfki.hu/igraph/) دارای قابلیت صادرات گراف به عنوان فایل های متنی ساده هستند. Cytoscape می تواند این فایل های متنی را بخواند و از آنها شبکه ها را بسازد. برای جزئیات بیشتر، لطفا بخشی از جداول واردات Free-Format را از بخش Networks Creating بخوانید.
آماده سازی داده ها برای ورود در cytoscape
برای ورود داده ها ابتدا باید جدولی شبیه به جدول زیر در اکسل تهیه مینماییم. در تهیه اکسل حتما در خانه A1 کلمه source و حتما در خانه B1 کلمه target را وارد نمایید. هر یال گراف دارای دو گره می باشد، پس در ستون اول یا source نام گره اول و در ستون target نام گره دوم را که با گره اول ارتباط دارد را وارد نمایید. سپس از طرق منوی file و سپس save as فایل اکسل را به صورت text tab delimited ذخیر نمایید.
|
البته همین دو ستون کافی است ولی برای حالت پیشرفته تر می توانید ستون های بیشتری مانند جدول زیر تولید کنید. برای اطلاعات بیشتر به لینک زیر مراجعه کنید.
http://manual.cytoscape.org/en/stable/Creating_Networks.html
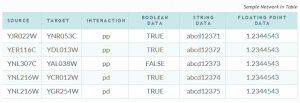
ستون اول: source که نام گره یا نود اول را نشان میدهد.
ستون دوم: target که نام گره یا نود دوم که با گره اول ارتباط دارد را نشان میدهد.
ستون سوم: نوع ارتباط
ستون چهارم: جهت دار بودن ارتباط نشان میدهد که این فیلد فقط دو حالت True و False را به منظور نشان دادن جهت دار یا بدون جهت بودن یال میپذیرد
ستون پنجم: نام نمادین گره اول
ستون ششم: نام نمادین گره دوم
وارد کردن اطلاعات در cytoscape
برای وارد کردن شبکه از جداول متنی / اکسل، لطفا این مراحل را دنبال کنید:
- File → Import → Network → File … را انتخاب کنید.
- یک فایل جدول (قبل) در پنجره محاوره انتخاب فایل انتخاب کنید.
- پارامترهای گراف راا مشخص کنید. اینکه کدام ستون ها از داده ها شامل منبع، هدف و نوع تعامل است. با کلیک بر روی هر سرستون انتخاب کنید.
- فعال کردن / غیرفعال کردن ستون های جدول: شما می توانید اطلاعات ستون را با فعال کردن نماد مربوطه در ویرایشگر ستون فعال / غیرفعال کنید.
-
روی دکمه OK کلیک کنید.
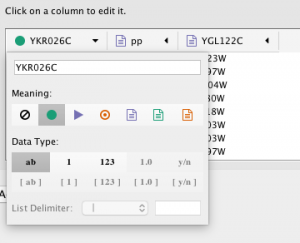
وراد کردن گراف شبکه های از پایگاه داده های عمومی
Cytoscape ویژگی ای به نام Network Import از پایگاه داده های عمومی دارد. کاربران می توانند از طریق این تابع به انواع مختلف پایگاه های داده دسترسی پیدا کنند.
در قسمت File → Import → Network → Public Databases ….
نوار جستجوی جدید در Cytoscape 3.6 در بالای پانل شبکه در کنترل پنل موجود است. از نوار جستجو، شما می توانید دسترسی به چندین پایگاه داده عمومی داشته باشید.
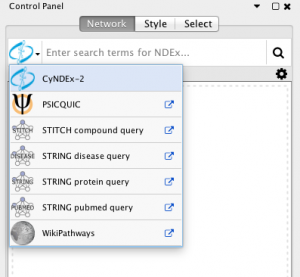
استفاده از وب سرویس در cytoscape
سرویس وب یک مکانیسم مستقل، مستقل از پلتفرم برای رایانه ها است تا بتواند از طریق اینترنت ارتباط برقرار کند. امروزه بسیاری از پایگاه های مهم بیولوژیکی اطلاعات خود را با API سرویس وب منتشر می کنند:
- فهرست وب سرویس های بیولوژیکی: http://taverna.sourceforge.net/services
- خدمات وب در EBI: http://www.ebi.ac.uk/Tools/webservices/
- تیم توسعه دهنده اصلی Cytoscape چندین مشتری خدمات وب را با استفاده از این چارچوب توسعه داده است. Cytoscape از بسیاری از خدمات وب پشتیبانی می کند از جمله:
- PSICQUIC: سرویس وب استاندارد برای مجموعه داده های تعاملی بیولوژیکی. فهرست کامل پایگاه های سازگار با PSICQUIC در اینجا موجود است. PSICQUIC از نوار جستجو در دسترس است.
- Pathway Commons: داده های یکپارچه از مسیر و منابع شبکه. لیست کامل منابع مورد نظر در اینجا موجود است. Pathway Commons از File → Import → Network → Databases عمومی در دسترس است ….
- STITCH و STRING: STITCH پایگاهی از تعاملات شناخته شده و پیش بینی شده بین مواد شیمیایی و پروتئین است. STRING یک پایگاه داده ای از تعاملات پروتئین و پروتئینی شناخته شده و پیش بینی شده است. STITCH و STRING از نوار جستجو در دسترس هستند و از File → Import → Network → Databases عمومی ….
- NDEx: تبادل اطلاعات شبکه (NDEx) یک چارچوب منبع باز فراهم می کند که دانشمندان و سازمان ها می توانند دانش شبکه بیولوژیکی را به اشتراک گذاشته، ذخیره، دستکاری و انتشار می کنند. NDEx از نوار جستجو در دسترس است. بخش های زیر نحوه وارد کردن شبکه از پایگاه های خارجی را توضیح می دهد. برای اطلاعات بیشتر لینک زیر را مطلعه کنید.
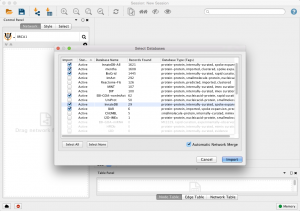
در ادامه نحوه وارد کردن شبکه از پایگاه های خارجی را توضیح می دهد. مثال: بازیابی شبکه های متقابل پروتئین-پروتئین از پایگاه های داده های چندگانه. در نوار جستجو، PSICQUIC را از منوی کشویی انتخاب کنید و یک یا چند عبارت جهت جستجو مانند BRCA1 تایپ کنید. برای شروع روی دکمه جستجو کلیک کنید. سپس پایگاه های داده مورد ظر خود را انتخاب کنید. و بعد برای وارد کردن داده های انتخاب شده شبکه روی دکمه import کلیک کنید.

این آموزش در روزهای آتی تکمیل خواهد شد
منابع:
[1] http://js.cytoscape.org/#performance/background
[2] http://en.wikipedia.org/wiki/Cytoscape#Usage
http://opentutorials.cgl.ucsf.edu/index.php/Tutorial:Introduction_to_Cytoscape
http://manual.cytoscape.org/en/stable/Creating_Networks.html
http://manual.cytoscape.org/en/stable/Creating_Networks.html
آدرس کانال تلگرام سایت بیگ دیتا:
آدرس کانال سروش ما:
https://sapp.ir/bigdata_channel
جهت دیدن سرفصل های دوره های آموزشی بر روی اینجا کلیک کنید.
جهت ثبت نام در دوره های آموزشی بر روی اینجا کلیک کنید.
بازدیدها: 8011
برچسبCytoscape آموزش CytoScape سایتو اسکیپ سایتواسکیپ معرفی cytoscape
همچنین ببینید
تشخیص موتیف یا زیر گراف های پرتکرار با برنامه Cytoscape
معرفی موتیف در مطالب قبلی به آموزش Cytoscape پرداختیم در آموزش امروز به نحوه تشخیص …
 خانه بیگ دیتا تحلیل شبکه های اجتماعی، متن کاوی، داده کاوی، اوسینت و داده های حجیم
خانه بیگ دیتا تحلیل شبکه های اجتماعی، متن کاوی، داده کاوی، اوسینت و داده های حجیم